人舌鳞癌细胞Tca8113、TSCCa等口腔癌细胞株的建立和应用,为研究口腔恶性肿瘤的生物学特性、诊断、治疗提供了有形的载体,为相关的放疗、化疗、基因治疗等方面的临床研究提供了巨大帮助。然而,随着细胞株的不断使用,Tca8113、TSCCa逐渐被HELA污染。
来源
Tca8113细胞由上海第二医科大学附属第九人民医院口腔颌面外科肿瘤生物实验室于1981年建系。初建系时细胞生长稳定、群体倍增时间为38.8h,分裂指数高峰值为61‰,平板集落形成率为86.0%;染色体多数稳定在66条,为亚三倍体;接种于C57BL和ICR小鼠皮下,成瘤率为100%。TSCCa细胞是由湖北医科大学口腔医学院建立,细胞常规培养在 RPMI +15%小牛血清的培养液中。
被HELA污染
2015年Fang Ye等人在FASEB J.发表的文章(PMID: 26116706)指出:在对国内113个来源单位的380株细胞进行STR分型时发现,许多国内建株的细胞系的STR图谱与HELA一致或高度相似,这其中就包括Tca8113和TSCCa这两株人舌鳞癌细胞系。
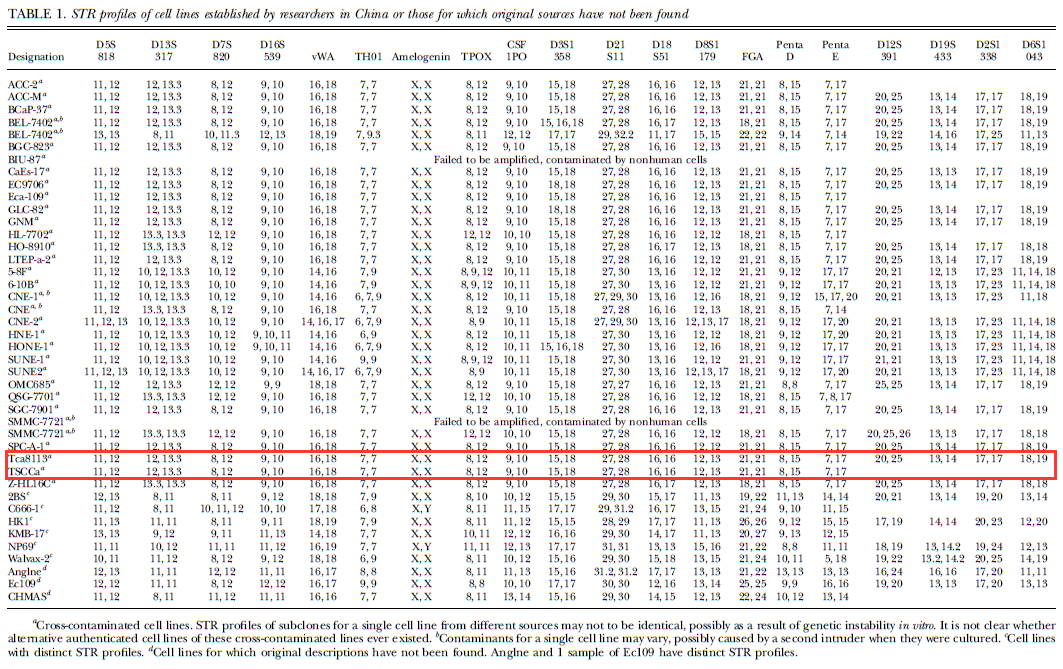
2017年Xiaocui Bian等人在Sci Rep.发表的文章(PMID: 28851942)进一步证实了Tca8113被HELA污染;同年,Yaqing Huang等人在PLoS One.发表的文章(PMID:28107433)也证实了TSCCa同样被hela污染。

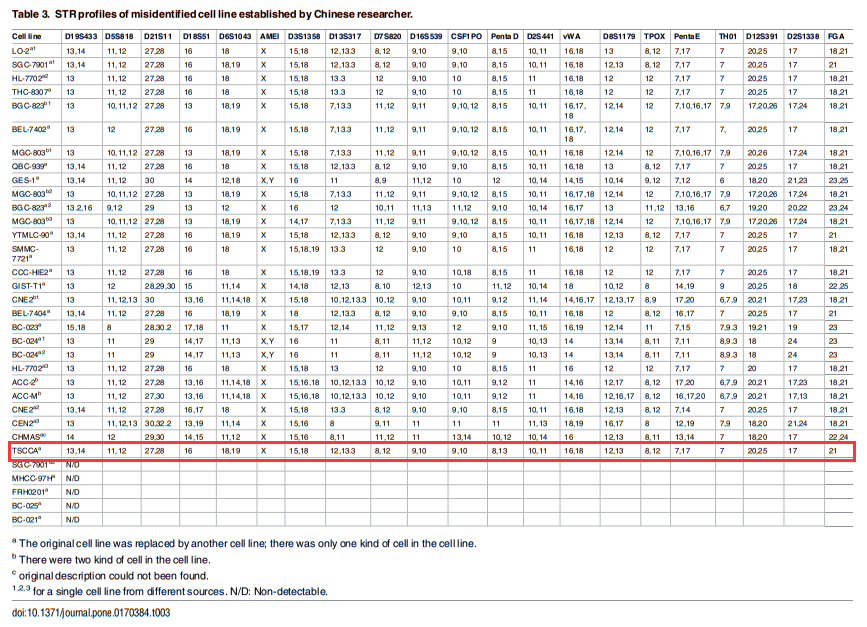
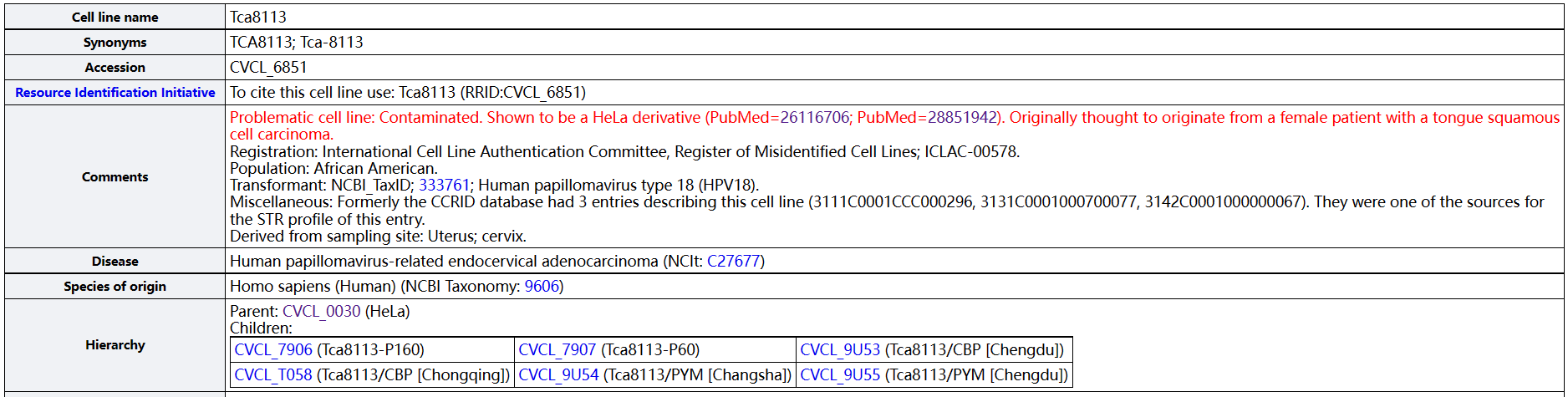
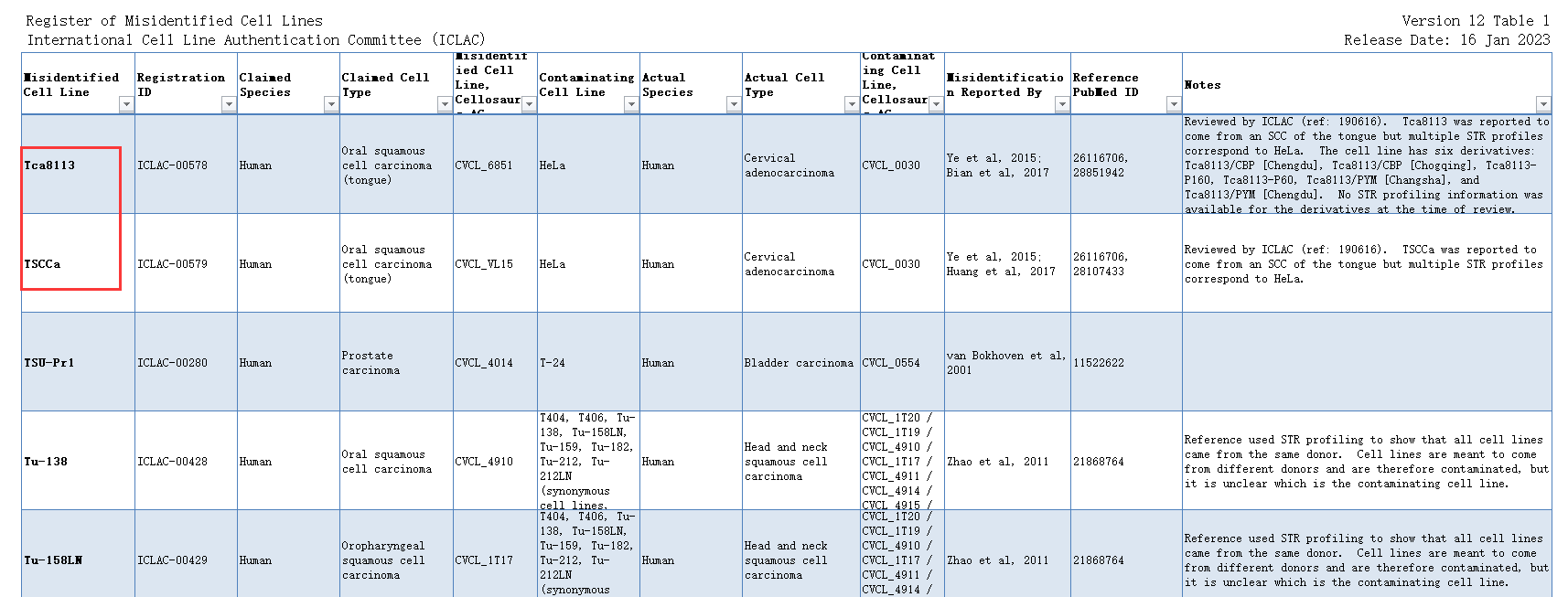
ExPASy数据库对这两株人舌鳞癌细胞系,已经做出“Problematic cell line: Contaminated”的标注。ICLAC的Register of Misidentified Cell Lines也将Tca8113和TSCCa收入其中。

参考文献:
1. www.jsdna.org/cell
2. http://www.jsdna.org/mobile/information/xuz
3. Fang Ye et al., Genetic profiling reveals an alarming rate of cross-contamination among human cell lines used in China. FASEB J. 2015 Oct;29(10):4268-72.
4.Huang Y et al., Investigation of Cross-Contamination and Misidentification of 278 Widely Used Tumor Cell Lines. PLoS One. 2017 Jan 20;12(1):e0170384.
5.Bian X et al., A Combination of Species Identification and STR Profiling Identifies Cross-contaminated Cells from 482 Human Tumor Cell Lines. Sci Rep. 2017 Aug 29;7(1):9774.
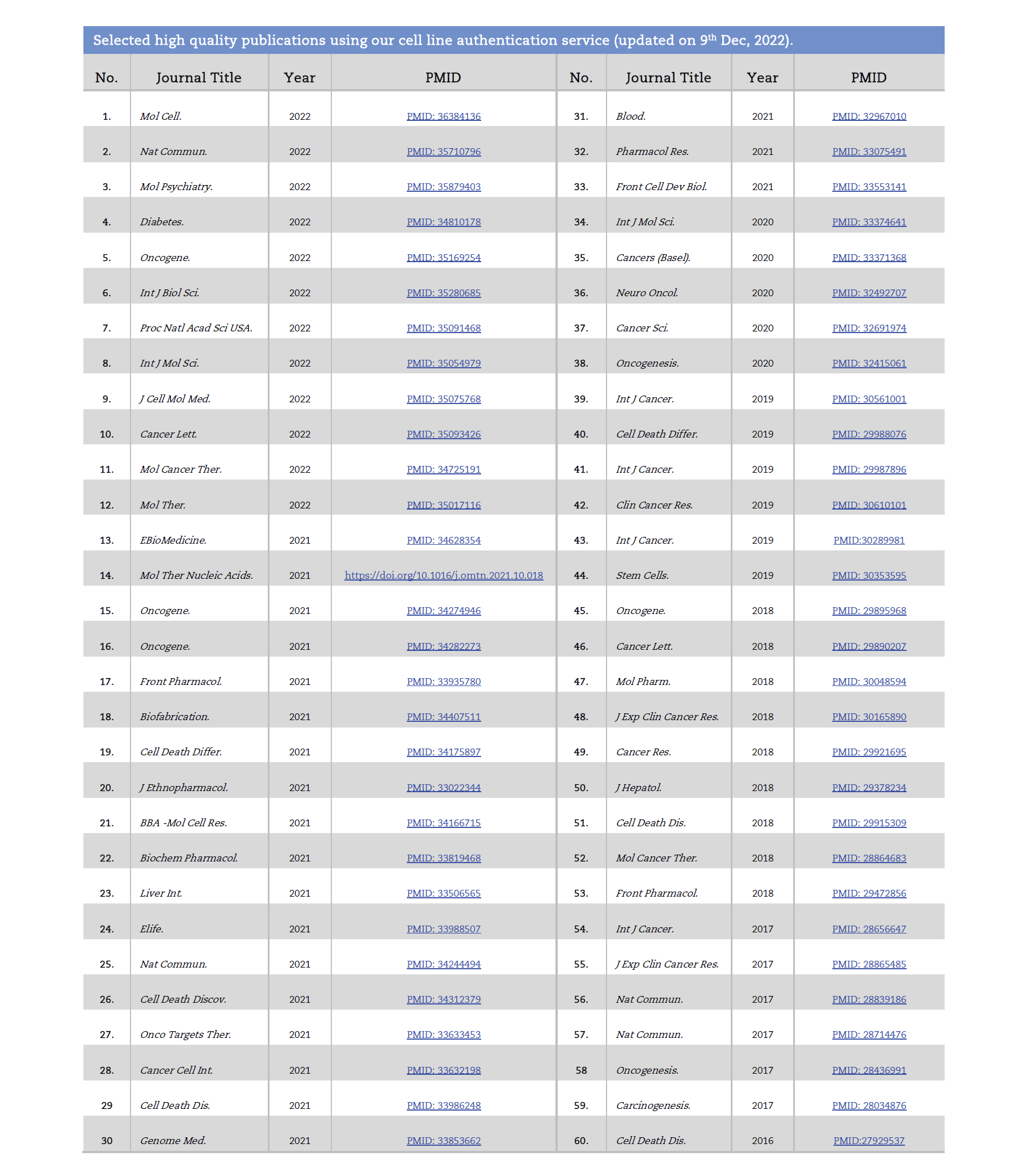
苏州鉴达(Genetic Testing Biotechnology, GTB)采用STR分型技术,针对人源细胞23个基因座、小鼠源细胞19个基因座(18个小鼠源基因座+1个人源基因座TH01)进行检测,3130XL型遗传分析仪进行毛细管电泳,与DSMZ、Cellosaurus、ATCC等数据库进行比对,出具权威英文报告,为全国近400家科研机构(高校、研究所、医院、公司)提供高质量服务,多家单位已利用鉴达细胞株鉴定服务在高水平期刊(如Clinical Cancer Research、Nature Commnications、Int J Cancer等)发表论文。
如需了解更多鉴达生物(Genetic Testing Biotechnology, GTB)相关细胞株鉴定信息,可扫码关注官方公众号。将本文转发至朋友圈(至少保留三天)。添加微信或QQ客服后,将朋友圈截图发给微信或QQ客服,并备注单位及课题组(示例:XX大学XX课题组)。后期可有两次机会享七五折优惠,原价1200元/株。微信:18112556907、QQ:2791427499
标注优惠政策:如客户在发表的高水平SCI论文(IF>10)中标注细胞株鉴定由我公司完成,我们承诺为客户免费检测两株细胞株。格式如下:The cell lines were characterized by Genetic Testing Biotechnology Corporation (Suzhou,China) using short tandem repeat (STR) markers.
Selected high quality publications using our cell line authentication